新闻网讯(通讯员钱开宇)2024年5月9日,武汉大学中南医院王行环教授团队在Nature Biotechnology(中科院一区Top,五年IF=59.1)发表题为Tracking single cell evolution using clock-like chromatin accessibility loci的研究论文。该研究为武汉大学首次以第一作者单位和第一通讯单位在Nature Biotechnology发表的研究型长文。

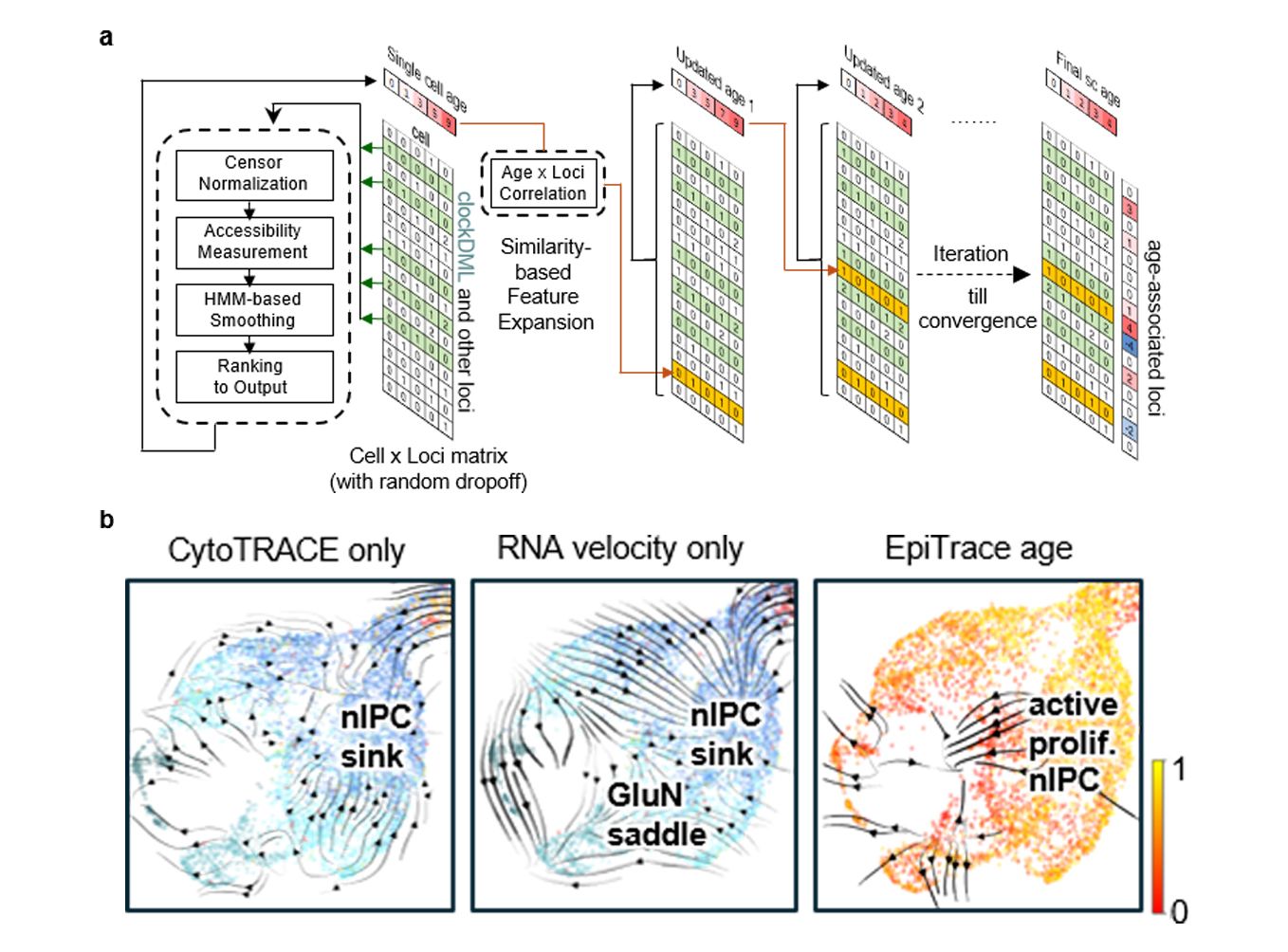
目前,对单细胞数据进行时间序列分析通常需要采用特定的实验手段,例如使用化学标记、高深度体细胞突变测序、谱系特异性反向遗传标记、多时点采样、单细胞甲基化测序等。为了便于从任意高通量单细胞测序数据集中重建精确的发育轨迹,需要找到数据中内涵的可用于报告细胞生成时间的内在生物标志物。课题组与北京优乐复生张翼教授团队自主研发了一种基于时钟样位点染色质可及性推断单细胞复制年龄的溯源技术(EpiTrace),实现了对单细胞染色质可及性(ATAC-seq)数据集的细胞年龄推断,为这一类单细胞测序数据集提供了时间参考系。EpiTrace与RNA速率、突变追踪、DNA甲基化年龄推断、多时点采样等技术之间具有良好的互补性。课题组使用EpiTrace追踪了膀胱癌发展过程中的关键节点,即TM4SF1阳性的癌细胞亚群(TPCS),发现了膀胱肿瘤发展的关键驱动因素。此外,课题组还将EpiTrace应用到追踪免疫治疗相关T细胞、肾脏单细胞、胶质母细胞瘤等,从溯源角度追踪了这些细胞的演化轨迹。

鉴于EpiTrace的重要性,Nature Biotechnology于本成果发表的同日,同期发表了题为Decoding cell replicational age from single-cell ATAC-seq data的Research Briefing(研究评述),汇聚了国际专家和编辑团队对本成果的评价和推介。
据悉,Nature Biotechnology杂志为研究方法学的顶级期刊,其刊发的科研成果通常都是生物医学领域开创性研究方法的重大革新。该研究得到了国家自然科学基金、中央高校基本科研基金、武汉大学中南医院的科研经费支持。
原文链接:https://www.nature.com/articles/s41587-024-02241-z
(编辑:赵冀帆)